Regulación de la transcripción
La información genética de una célula está contenida en su DNA. El DNA contiene la información necesaria para crear miles de moléculas diferentes y con ello conforma un individuo completo. Un organismo procarionte como una bacteria, consta de millones de nucleótidos, en tanto que uno eucarionte, como el humano, cuenta con varios billones. A pesar de contener toda esa información, la célula expresa sólo una fracción de sus genes, lo que le da un fenotipo característico, de tal modo que cada uno de los 10 000 tipos de células que constituyen a los organismos  multicelulares expresan genes diferentes. Aunque los diversos tipos de células en un organismo multicelular contienen la misma información genética, difieren ampliamente en estructura y función. Una neurona y un linfocito por ejemplo son muy diferentes, por lo que sería difícil pensar que estas dos células contienen el mismo genoma como sucede en realidad. Por esta razón, se pensaba que los genes se perdían cuando las células se diferenciaban. Ahora se sabe que las células de un organismo multicelular son distintas porque sintetizan diferentes RNAm y por ende diferentes proteínas a partir del mismo genoma.
multicelulares expresan genes diferentes. Aunque los diversos tipos de células en un organismo multicelular contienen la misma información genética, difieren ampliamente en estructura y función. Una neurona y un linfocito por ejemplo son muy diferentes, por lo que sería difícil pensar que estas dos células contienen el mismo genoma como sucede en realidad. Por esta razón, se pensaba que los genes se perdían cuando las células se diferenciaban. Ahora se sabe que las células de un organismo multicelular son distintas porque sintetizan diferentes RNAm y por ende diferentes proteínas a partir del mismo genoma.
REGULACIÓN DE LA EXPRESIÓN GÉNICA
Uno de los principios fundamentales de la biología celular
es que la actividad y propiedades de cada célula están
determinadas por las proteínas que contienen. Pero,
¿qué determina los tipos y la cantidad de proteínas que
se encuentran en una célula? ¿Qué determina la frecuencia
con que los RNAm son traducidos y la estabilidad de
las proteínas?
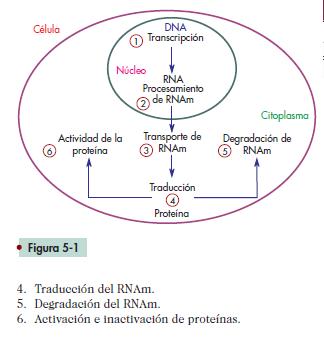
El término expresión génica se refi ere al proceso
mediante el cual la información codifi cada en un gen es
traducida en una proteína. Aunque la expresión de un
gen puede regularse a diferentes niveles desde su transcripción
a partir del DNA, hasta la formación de una
proteína madura y activa (fi g. 5-1), el mayor punto de
control se da a nivel de la transcripción. Sin embargo, la
expresión génica puede ser regulada en cualesquiera de
los siguientes niveles:
1. Transcripción.
2. Procesamiento del transcrito primario de RNA.
3. Transporte del RNAm al citoplasma.
CONTROL TRANSCRIPCIONAL
La primera evidencia de que había proteínas reguladoras
fue descubierta en 1950. Al realizar análisis genéticos
en bacterias, se observó que ciertas proteínas se unían
a secuencias específi cas del DNA y que esta unión era
necesaria para la activación e inactivación de genes.
Control transcripcional en procariontes
En las bacterias, el control de la transcripción está dado
principalmente por los cambios en el ambiente donde se
encuentran, lo que determina su crecimiento y proliferación.
Aunque en organismos multicelulares la expresión
de algunos de sus genes es también controlada por
la disponibilidad de alimento, en la mayoría de ellos el
proceso de la transcripción es regulado por programas
genéticos que controlan su desarrollo embrionario y la
diferenciación celular. Muchos de los principios de regulación
transcripcional se descubrieron en las bacterias y
se aplican también a células eucariontes.
.
Control transcripcional en eucariontes
La transcripción génica es controlada por una región de
DNA ubicada cerca del sitio de inicio de la transcripción
a la que se le llama región reguladora. Algunas regiones
reguladoras son sencillas y constan de un fragmento
pequeño de DNA de secuencia defi nida; estas regiones
están regidas por una sola señal y actúan controlando la
actividad de los genes. Otras son complejas, responden a
una variedad de señales y actúan como pequeños microprocesadores
que interpretan e integran el “encendido
y apagado” de genes vecinos. La transcripción es controlada
por proteínas especializadas llamadas proteínas
reguladoras de un gen o factores transcripcionales.
Un mismo factor transcripcional puede servir como controlador
común a muchos genes al mismo tiempo. Esto
se debe a que todos ellos comparten la misma secuencia
en su promotor, la cual es reconocida por el factor transcripcional.
Las proteínas reguladoras se unen a secuencias específi
cas de DNA de aproximadamente 20 pares de nucleótidos
de longitud cerca de los genes que regulan. El
reconocimiento entre la proteína y el DNA se realiza a
través del surco mayor y se debe a que la estructura de
la proteína es complementaria a la superfi cie espacial
de la doble hélice; la proteína hace contacto con el DNA
Activación e inactivación de genes
La regulación de la transcripción en eucariontes es muy
compleja y difi ere de la de procariontes, principalmente
en los siguientes pasos:
a) La polimerasa del RNA eucarionte no puede iniciar
por sí misma la transcripción; requiere de un grupo de
proteínas reguladoras llamadas factores generales de
la transcripción, las cuales se ensamblan al DNA en
un sitio llamado promotor, localizado corriente arriba
del sitio de inicio de la transcripción. El ensamblaje
de los factores generales consta de varias etapas en
donde la velocidad depende de las señales reguladoras
que se reciban.
b) La mayoría de los factores transcripcionales actúa a
miles de bases de distancia del promotor que regulan,
lo que signifi ca que un promotor puede ser controlado
por un número ilimitado de secuencias reguladoras
localizadas a lo largo de la cadena del DNA.
Factores generales de transcripción
El hallazgo de que la polimerasa del RNA eucarionte por
sí sola no puede iniciar la transcripción condujo al descubrimiento
de proteínas adicionales conocidas como
factores generales de la transcripción.
Estas proteínas, conocidas como TFIIA, TFIIB, TFIID,
TFIIE, TFIIF, TFIIH y TFIIJ (por sus siglas en inglés transcriptional
factor, TF y II por estar asociadas a la polimerasa
del RNA II), se ensamblan en el promotor de los
genes de manera coordinada asistiendo la unión y acción
de la polimerasa del RNA. El ensamblaje se inicia con
la unión del factor transcripcional TFIID a la secuencia
TATA en el promotor (formada por dos tipos de proteínas
diferentes: TBP y TAF, TATA binding protein y TBP asociates
factors, respectivamente). Después se incorporan
los factores TFIIA y TFIIB y sólo entonces la polimerasa
de RNA II acomplejada con el TFIIF puede unirse al promotor.
TFIIE, TFIIH y TFIIJ se acoplan después a este
complejo. Una vez reunidos todos estos elementos, TFIIE
con su acción de helicasa desenrolla el DNA y TFIIH
EDICIÓN DEL RNA
Este tipo de control postranscripcional se refi ere a la
modifi cación debida a inserciones o eliminaciones de
nucleótidos en la secuencia original del RNAm, provocando
cambios en el mensaje original. La modifi cación
más frecuente es la inserción del uracilo, con lo que se
altera hasta 50% la secuencia original.
En mitocondrias de plantas, se han observado cambios
de citocinas por uracilo (C a U) sin inserción o eliminación
de nucleótidos, afectando sólo 10% de la secuencia
de aminoácidos de las proteínas formadas a partir
de estos RNAm. El descubrimiento de este mecanismo de
edición ha permitido sugerir que es la herramienta más
utilizada por la célula para cambiar la expresión de los
genes.
El proceso de edición del RNAm es limitado en mamíferos
y sólo se ha observado en los genes de la ApoB y
de la proteína de canal del calcio en el cerebro. En el
primer caso, una citocina se cambia por un uracilo, generando
un codón de terminación prematuro que produce
una versión truncada de la proteína; en el segundo caso,
se cambia un nucleótido a la mitad de la molécula del
RNAm. Esto origina el reemplazo de un aminoácido por
otro, lo que altera la permeabilidad del canal del calcio.
Control del transporte de RNA
Transporte del núcleo al citoplasma
El RNA transcrito primario es aproximadamente 10 veces
más grande que el RNAm maduro que sale al citoplasma.
Se estima que sólo 20% de la longitud del RNA sintetizado
sale del núcleo; el resto (80%) es degradado en el núcleo
antes de salir. El RNA como cualquier otra molécula sale
del núcleo a través del poro nuclear; se trata de un proceso
activo que requiere de un nucleótido modifi cado que
sirve como cubierta en el extremo 5′ de la molécula y de
la adición de una cadena de adeninas en el extremo 3′
(poliadenilación) llamada cola de poli A.
Cuando una molécula de RNAm recién sintetizada
cruza por un poro nuclear y se introduce al citoplasma,
se encuentra con los ribosomas, los cuales la traducen en
una cadena polipeptídica. Si el RNAm codifi ca para una
proteína de secreción o de membrana, la presencia de un
péptido, señal en la región aminoterminal determina su
transporte hacia el retículo endoplásmico. Este péptido
es reconocido por la célula tan pronto sale del ribosoma
y entonces el complejo formado por el ribosoma, el
RNAm y la proteína naciente se dirigen a la membrana
del retículo endoplásmico, donde la cadena polipeptídica
terminará de sintetizarse. En otros casos, la proteína
entera es sintetizada por ribosomas libres en el citosol, y
señales en su estructura la dirigen al sitio en la célula en
donde se necesita. Asimismo, algunos RNAm se dirigen a
ciertos organelos para sintetizar ahí las proteínas propias
del mismo.
Control de la traducción
Sitio de inicio
La traducción comienza cuando la subunidad pequeña
del ribosoma reconoce el codón de iniciación en el
RNAm. Los nucleótidos vecinos participan en este reconocimiento.
Si éste es defi ciente, la subunidad ribosómica
Cuadro 5-1 Modifi caciones postranscripcionales
Atenuación de la transcripción Terminación prematura de la transcripción, ocasionada por interacción con la
polimerasa del RNA
Control del procesamiento del RNA Retiro de intrones del RNAm precursor; incluye también el corte y empalme
alternativo
Edición del RNA corte y empalme Inserción o eliminación de nucleótidos del RNAm; la modificación más frecuente es la
adición de un uracilo
Control del transporte del RNA El paso del RNAm por el poro nuclear al citoplasma implica que se ha modificado
adecuadamente
Control de la traducción El reconocimiento del codón de inicio de la traducción puede ser deficiente
generando orígenes alternativos
Fosforilación en la síntesis de proteínas El factor de iniciación de la traducción del IF2 es regulado por fosforilaciones
Proteínas inhibidoras de la traducción Proteínas que se unen al extremo 5’ del RNAm bloqueando la traducción
Control de la degradación del DNA Colas de poli A menores a 30 Nt inducen la degradación del RNA
Mecanismos de fosforilación en la síntesis de proteínas 69
ignorará el primer codón AUG y saltará hasta el segundo
o tercero. Este fenómeno, conocido como búsqueda de
escape, es una estrategia para producir dos o más proteínas,
diferentes en su extremo aminoterminal a partir de
un mismo RNAm.
En RNAm virales, se realiza la traducción usando este
tipo de mecanismos. Estos RNAm cuentan con secuencias
de nucleótidos específi cas llamadas sitios internos,
las cuales no son reconocidas por el ribosoma, y la traducción
se inicia en el segundo codón AUG.
MECANISMOS DE FOSFORILACIÓN
EN LA SÍNTESIS DE PROTEÍNAS
Otro mecanismo por el cual se puede regular la expresión
de un gen es mediante fosforilaciones. Las células eucariontes
disminuyen la síntesis de proteínas en condiciones
adversas (p. ej., carencia de factores de crecimiento,
infecciones víricas, choque térmico, etc.). En este mecanismo
de regulación, participa el factor de iniciación
EIF-2 (elongation increasing factor, EIF), el cual es fosforilado
por cinasas específi cas, con lo cual se disminuye
la síntesis de proteínas. El EIF-2 forma un complejo con
el GTP y media la unión del metionil-RNAt a la subunidad
ribosómica pequeña, facilitando el reconocimiento
del codón de iniciación. El EIF-2 hidroliza GTP y se libera
de la subunidad ribosómica pequeña. Sólo entonces
la subunidad ribosómica grande se acopla para formar el
ribosoma e iniciar la síntesis de proteínas.
Debido a que el EIF-2 se fi ja fuertemente a GDP, se
requiere otra proteína denominada EIF-2B para liberar
a GDP; otra molécula de GTP puede entonces unirse de
nuevo a la molécula del EIF-2. La fosforilación del EIF-2
bloquea el recambio de GTP; ya fosforilado el EIF-2 se
une con mayor fuerza al EIF-2B. Se esperaría que la disminución
de la actividad del EIF-2 mermara la traducción
de todos los RNAm en la misma proporción. Contrario
a esto, la fosforilación del EIF-2 tiene efectos selectivos,
incrementado la traducción del RNAm específi cos. La
regulación por el EIF-2 es importante en el paso de mitosis
a la fase Go en células de mamíferos en la cual se
minimiza la síntesis de proteínas.
Proteínas inhibidoras de la traducción
La traducción puede ser bloqueada también por proteínas
que se unen a los RNAm cerca del extremo 5′, donde
se inicia la traducción. Este tipo de mecanismo se llama
control negativo de la traducción. El acoplamiento de
tales proteínas a una molécula de RNAm disminuye o
inhibe la traducción.
Los RNAm en células bacterianas son muy inestables;
su vida media es de unos minutos. Debido a esto, una
bacteria puede adaptarse rápido a cambios ambientales.
En células eucariontes, el RNAm es más estable; por
ejemplo, el de la globina β tiene una vida media de 10 h,
cuando la de otros es de sólo 30 min. Los más inestables
codifi can a menudo para proteínas reguladoras, cuya síntesis
cambia rápidamente ante un estímulo. La inestabilidad
de estos RNAm se debe a que su secuencia es rica en
A y U en la región 3′ no traducida (UTR), lo que acelera
la degradación del RNAm por estimulación en la eliminación
de la cola de poli A. Otros RNAm inestables contienen
sitios de reconocimiento en sus extremos 3′ UTR
para endonucleasas que cortan al RNAm. Sin embargo,
la estabilidad de un RNAm puede cambiar en respuesta a
señales extracelulares. En el caso del RNAm que codifi ca
para las histonas, por ejemplo en la fase de síntesis (S)
del DNA en el ciclo celular, periodo en el que se requiere
de nuevas histonas, tiene una vida media de 1 h; cuando
la síntesis de DNA se detiene, los RNAm se inestabilizan
y son degradados en pocos minutos. Así, si la síntesis de
DNA se inhibe con algún fármaco, los RNAm de las histonas
se inestabilizan debido quizá a la acumulación de
histonas libres en ausencia de nuevos DNA. La velocidad
de degradación de los RNAm depende de manera directa de
las señales que actúan en el extremo 3′, en donde se sospecha
que comienza la degradación del RNAm.
Control de degradación del RNAm
Cola de poli A y estabilidad del RNAm
La adición de la cola de poli A a una molécula de RNAm
sucede en todos los RNAm de eucariontes, en el núcleo.
En el citosol, la cola de poli A, con una longitud promedio
de 200 nucleótidos, se va acortando con el tiempo. No se
observan colas de menos de 30 adeninas, lo que sugiere
que este es el tamaño mínimo requerido para mantener
la estabilidad del RNAm. La observación de que RNAm
con colas de sólo 10 a 30 adeninas no son traducidas apoya
esta aseveración.
Interrupción de la traducción
El proceso de síntesis de proteínas es automático; esto es,
una vez iniciado debe terminarse. En casos especiales,
un proceso llamado recodifi cación traduccional puede
alterar el curso fi nal de la síntesis de una proteína. Los
tipos de recodifi cación más observados a menudo son los
cambios en el marco de lectura. Este tipo de recodifi cación
se usa de ordinario por retrovirus, en el que más de
una proteína se sintetiza a partir de un solo RNAm. Estos
virus producen tanto las proteínas de la cápside (proteínas
gag), como la transcriptasa inversa vírica y las integrasas
(polimerasas de proteínas) del mismo transcrito.
Los virus necesitan muchas más copias de las proteínas
gag que de las polimerasas de proteínas. Para cumplir
con estas demandas, los virus hacen este ajuste cuanti70
Capítulo 5 Regulación de la transcripción
tativo al tener los genes gag y pol en marcos de lectura
abierta diferentes, asegurando con un codón de terminación
en el extremo de la secuencia codifi cadora para gag,
que sólo traduzca esta proteína.
Todos los mecanismos de control postranscripcionales
dependen del tipo de molécula de RNA, por lo que algunos
pueden ser controlados a través del procesamiento,
edición o degradación del RNAm. Dichos mecanismos se
acompañan a menudo de proteínas de unión al RNA especializadas.
En otros casos, se debe a la presencia de moléculas
de RNA complementarias al RNAm que al unirse a
él bloquean su transcripción. A este tipo de estrategias se
le conoce como RNA antisentido y se han empleado de
modo experimental para regular la expresión de algunos
genes de células procariontes y eucariontes. Los estudios
con RNA antisentido son de gran interés, ya que nos
permiten entender procesos evolutivos. Se cree que las
primeras células carecían de DNA y proteínas, y sólo contenían
RNA. Estas células primitivas utilizaban el mecanismo
antisentido para regular sus funciones.
Una buena parte de las reacciones catalizadas por
RNA que suceden en las células hoy en día representan
los descendientes de moléculas fósiles que controlaban el
metabolismo celular hace más de 3.5 billones de años. En
la actualidad, gracias a la tecnología del DNA recombinante
se pueden obtener in vitro grandes cantidades de RNA
puro de cualquier tipo que, en presencia de polimerasas
de RNA, nos permiten estudiar con detalle las reacciones
químicas catalizadas por RNA. A través de estos y más
estudios, se pretende conocer en un futuro no muy lejano
los mecanismos de regulación de la expresión génica de
las células in vivo.